El primer pangenoma humano revela 120 millones de letras más en el ADN
Un consorcio internacional publica un borrador genético de la humanidad que por primera vez incluye la diversidad de la especie, lo que facilitará el tratamiento de enfermedades como el cáncer

Un hombre abrió un periódico el 23 de marzo de 1997 y cambió el mundo para siempre. En una de las páginas del diario estadounidense The Buffalo News había un anuncio llamativo: “Se busca a 20 voluntarios para participar en el Proyecto Genoma Humano. [...] El resultado tendrá una enorme repercusión en el futuro progreso de la medicina”. Aquel lector acudió a la llamada, donó unos mililitros de sangre y se incorporó a un proyecto de 3.000 millones de dólares que desembocó en 2003 en el llamado genoma de referencia humano, compuesto al 70% por el ADN de aquel hombre, con retales de otras dos decenas de personas. Aquella información genética, efectivamente, cambió la historia de la humanidad, pero era insuficiente, al excluir la diversidad de la especie humana. Un consorcio internacional publica este miércoles una alternativa más sofisticada, elaborada con las secuencias genéticas de 47 personas de diferentes regiones del planeta. Es el primer borrador del denominado pangenoma humano.
El genoma de una persona, su ADN, es el manual de instrucciones presente en cada una de sus células. Es un texto de unos 3.055 millones de letras (ATGGCGAGT…), en el que cada letra es simplemente la inicial de un compuesto químico con diferentes cantidades de carbono, hidrógeno, oxígeno y nitrógeno. La G, por ejemplo, es la guanina: C₅H₅N₅O. El genoma de dos personas coincide al 99,9%, pero ese 0,1% restante son millones de letras que hacen único a un ser humano y que pueden esconder las claves de sus enfermedades. Si el genoma de referencia de 2003 es una secuencia lineal, el nuevo pangenoma humano se puede imaginar como un mapa de caminos en el que un genoma individual es un trayecto concreto, en palabras de Benedict Paten, biólogo computacional de la Universidad de California en Santa Cruz (Estados Unidos) y uno de los líderes de la investigación.
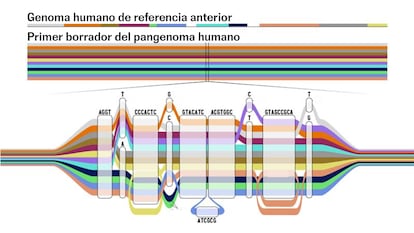
El borrador añade 119 millones de letras al modelo empleado hasta ahora. Los autores del trabajo, agrupados en el Consorcio Pangenoma Humano de Referencia, explican que la escasa diversidad del actual genoma de referencia ha provocado “un efecto farola”, un fenómeno que debe su nombre al chiste del borracho que está buscando las llaves de su casa en el suelo de una calle, de noche, bajo una farola encendida. Un policía lo intenta ayudar y, tras unos minutos de búsqueda infructuosa, el agente pregunta al vecino ebrio si está seguro de que ha perdido las llaves ahí. “No, se me han caído en el parque, pero es aquí donde hay luz”, responde el hombre. Los científicos llevan dos décadas buscando posibles variantes genéticas donde era más fácil mirar: dentro de los límites del genoma de referencia, que además de ignorar la diversidad humana, estaba lleno de agujeros por la falta de precisión de la tecnología.
Benedict Paten y sus colegas han trabajado durante años en desarrollar nuevas herramientas, capaces de leer el ADN con una exactitud sin precedentes, con apenas un error cada 200.000 letras. Varios miembros del equipo también han participado en el Consorcio T2T, que logró hace un año la primera secuencia verdaderamente completa de un genoma humano. Hasta entonces solo se había conseguido leer el 92%. El restante 8% era como las piezas de cielo azul en un puzle: demasiado repetitivas como para encontrar fácilmente su posición.
Una medicina “más justa”
La genetista Karen Miga, de la Universidad de California en Santa Cruz, proclamó el martes en una rueda de prensa que la diversidad del pangenoma inicia una nueva era, “más justa”, en la medicina. Los 47 genomas incorporados por el momento proceden sobre todo de África (24) y América (16), incluyendo cuatro peruanos de Lima, otros cuatro colombianos de Medellín y ocho puertorriqueños. Seis genomas son asiáticos y solo uno es de Europa, un continente que ya está sobrerrepresentado en las bases de datos genéticas. El objetivo del equipo es alcanzar los 350 genomas completos en un solo pangenoma, que se publicará a mediados del año 2024. El primer borrador se presenta este miércoles en la revista Nature.

El científico español Santiago Marco, que ha desarrollado algoritmos y herramientas de software para el pangenoma, explica la magnitud del desafío técnico. Las máquinas actuales no pueden leer un genoma del tirón, sino que leen miles de millones de pequeños fragmentos de forma aleatoria y repetida. “Ensamblar un genoma de una persona es como reconstruir un gran libro, con 3.000 millones de letras, poniendo en común párrafos y páginas desordenadas, como si de un gran puzle se tratara”, señala Marco, del Centro Nacional de Supercomputación, en Barcelona. “La construcción de un pangenoma de referencia puede requerir procesar 100 veces más información”, advierte.
El bioinformático Francisco Martínez Jiménez utiliza en su día a día el genoma de referencia, como modelo para buscar alteraciones específicas de los tumores de los pacientes del Vall d’Hebron Instituto de Oncología, en Barcelona. El especialista explica que, si los ancestros del enfermo son, por ejemplo, de Sudamérica, África o el sudeste asiático, es “mucho más difícil” detectar esas alteraciones, porque el genoma de referencia actual está elaborado principalmente con ADN de personas de origen europeo. “Que haya diversidad genética en el pangenoma es muy relevante, particularmente en el cáncer”, aplaude.
Martínez Jiménez ha encabezado el análisis del genoma completo de más de 7.000 tumores primarios y metastásicos, de 71 tipos de cáncer. Sus resultados, también publicados este miércoles en la revista Nature, muestran que en ciertos tipos de tumores, como los de próstata, tiroides y algunos de mama, las diferencias genéticas entre el cáncer primario y la metástasis son “muy importantes”, mientras que en otros, como el de páncreas, son sutiles. “Las metástasis per se no parecen estar explicadas, en general, por una alteración específica genómica, sino posiblemente por cambios en el microambiente del tumor, como una privación del sistema inmune en determinadas localizaciones o una mayor irrigación por vasos sanguíneos, con más nutrientes”, subraya el bioinformático, que hizo el trabajo en la Universidad de Utrecht, en Países Bajos.
El biólogo Benedict Paten insiste en que el pangenoma humano es de momento un borrador y pide paciencia hasta que se vea un auténtico impacto en la medicina. “Hay errores de ensamblaje —no demasiados, pero sí algunos— que sabíamos que cometeríamos y que queremos corregir”, admite. Otro de los coautores de la investigación, Erik Garrison, de la Universidad de Tennessee, ha mostrado su entusiasmo en un comunicado. En su opinión, el primer borrador del pangenoma humano “es tan emocionante e inesperado como las primeras observaciones de regiones desconocidas de nuestro propio planeta o del sistema solar, pero en este caso están tan cerca que literalmente definen nuestra naturaleza física”.
Puedes escribirnos a mansede@elpais.es o seguir a MATERIA en Facebook, Twitter, Instagram o suscribirte aquí a nuestro boletín.
Sobre la firma